2. 华东师范大学 计算机科学与软件工程学院, 上海 200062
2. School of Computer Science and Software Engineering, East China Normal University, Shanghai 200062, China
马尔科夫链被广泛应用到各个领域, 包括自动控制、经济、物理、生物等.它用于描述具有马尔科夫性质的离散事件随机过程.该过程中, 在给定知识或信息的情况下, 过去发生的事无关于对将来事情的预测. PRISM[1] (Probabilistic Model Checker)是一种基于马尔科夫链的模型验证工具, 它用于验证系统的相关性质, 如安全性、有用性等.同时, 它支持多种模型, 如离散时间马尔科夫链DTMC(Discrete-Time Markov Chain)、连续时间马尔科夫链CTMC(Continuous-Time Markov Chain)、马尔科夫决策过程MDP(Markov Decision Processes).在PRISM中, 当系统描述为DTMC或者是MDP时, PRISM所要求的系统属性输入语言为概率时序逻辑PCTL或PCTL*和LTL (Linear Temporal Logic); 当系统描述为CTMC时, 所需验证的系统所要验证的性质为概率时序逻辑PCTL(Probabilistic Temporal Logic).布尔网络由科学家Kauffman在1969年首次提出[2], 主要应用于基因调控网络, 它的用途广泛, 在系统生物学、物理学、临床医学、生理学等方面都有着重要的应用[3].对于表示基因调控网络的布尔网络来说, 找出吸引子在生物系统学和生物信息学上都有着关键的作用[4], 有学者认为布尔网络的吸引子可以反映细胞的类型, 这对理解细胞的动态平衡和肿瘤的发展提供了一个新的视角[5-6].因此, 有很多关于吸引子数目、长度等性质的研究[7-9].而找出吸引子是一个复杂的问题, 这一领域的主要研究方向在于如何降低找到吸引子的算法复杂度[4, 10].目前, 寻找吸引子的思路主要在于简化布尔网络.文献[11]中, 先把布尔网络转换为真值表, 后寻找真值表中的子序列, 根据吸引子的定义, 吸引子可以和真值表中连续的序列对应, 因此可以通过对真值表进行处理来简化吸引子的找寻流程.通过去除真值表中不连续的序列, 即无法到达吸引子的序列, 来降低算法复杂度.实验表明, 此法可以找到200个节点大小布尔网络的吸引子.还有在简化过的布尔网络的基础上, 改进算法, 使之适用于多进程并行计算吸引子, 但是此算法不适合用到太过大的布尔网络中, 只适用于数十个到数百个节点的布尔网络[12].本文用概率模型检测技术应用于布尔网络, 以此简便地找出吸引子, 简化找寻吸引子的过程, 提出了一个可以把布尔网络转换为离散时间马尔科夫链的方法, 通过增加基因扰动找到基因之间的相互关系[13].找到基因之间的相互关系可以帮助研究人员发现某些疾病的全新治疗角度, 比如对xCT基因的增强有助于脑血栓的治疗[14], 所以找到增强xCT基因表达的基因有助于脑血栓的治疗研究.目前, 对于基因间相互关系的研究主要在文本挖掘的方向上, 通过挖掘现有数据结合, 分析基因间相互关系.有使用文本挖掘技术miRTex找寻miRNA和gene-miRNA的相互调节关系, 这项技术全面处理了所有国际性综合生物医学信息书目数据库的所有开发信息集合, 并且达到了良好的精度[15];还有针对特定疾病的研究, 比如通过生物医学文本挖掘工具Chilibot对从Pub Med中所获相关文献的摘要进行分析, 通过对相互作用的深入分析, 发现了白血病和基因的相互作用关系[16].本文在将基因调控网络转换为离散时间马尔科夫链的基础上, 把两个问题合并为一个问题, 在同一模型检测工具PRISM下, 使用相似的方法一次处理两个不同的问题.本文以一个大鼠干细胞基因调控网络为例, 提出了从布尔网络到离散时间马尔科夫链的转换方法, 并且使用模型检测工具PRISM找到了在某个特定初始状态下的吸引子; 另外, 还利用增加基因扰动来找寻基因间相互关系的方法, 找到了某些大鼠干细胞基因的相互关系.
本文后续结构为:第1节介绍本文知识背景, 包括布尔网络、离散时间马尔科夫链以及模型检测工具PRISM; 第2节介绍模型转换, 将表示为基因调控网络的布尔网络转换为离散时间马尔科夫链; 第3节以生成的离散时间马尔科夫链为基础, 使用模型检测工具PRISM找到吸引子, 通过增加扰动找到基因之间的相互关系, 并对实验结果进行分析; 第4节进行总结和展望.
1 预备知识本节介绍布尔网络到离散时间马尔科夫链转换所需要的预备知识, 包括离散时间马尔科夫链与布尔网络的定义和性质、模型检测工具PRISM的用途和语法.
1.1 离散时间马尔科夫链离散时间马尔科夫链[17]是在一个状态空间内的一个随机状态转换的过程.一般地, 需要这个状态转换具备"无记忆性", 即下一个状态的性质只取决于当前状态的性质, 而跟这个状态之前所有的事件都无关, 这种"无记忆性"又被称为马尔科夫性质.因此, 离散时间马尔科夫链也指一个具有马尔科夫性质的随机过程所经过的一系列变量.在实际应用中, 离散时间马尔科夫链主要用于描述一系列相互关联事件的系统, 其中下一个事件只取决于当前事件而与历史事件无关.离散时间马尔科夫链被广泛用在很多统计模型中.一个离散时间马尔科夫链既可以为离散时间的模型建模, 又可以为概率选择模型建模, 方式是为不同状态的转换赋以不同速率.离散时间马尔科夫链的定义如下.
定义1 假设
DTMC模型中, 一个状态
布尔网络是一种有向图, 其中的节点代表系统的元素, 边代表元素之间的调控关系.每个节点表示一种基因的表达状态, 而每个节点中的每个基因则被刻画为激活(1)与抑制(0)两种状态.因此一个含有
定义2 一个布尔网络[19]是一个二元组,
在布尔网络中, 每个节点状态的变化将由其更新函数和当前节点的状态共同决定.通过这种更新方式网络会产生出一系列的动态节点变化.布尔网络的这种特点使得它在生物学中广泛被应用, 其中最常见的应用就是描述基因调控网络.基因
| $ \begin{align} X_i(t+1)=f_i(X_{j_1(i)}(t), X_{j_2(i)}(t), \cdots, X_{j_k(i)}(t)), \end{align} $ | (1) |
其中,
布尔网络中的状态通常分为两类:暂态(transient state)和吸引子(attractor).暂态指在整个运行过程系统仅经过一次的状态.吸引子是系统经过一段时间后不断重复经历的状态.简单来说, 暂态就是在状态更新的过程中不会循环出现的状态.吸引子则可以分为两类, 如果吸引子只包含一个状态, 称之为单吸引子(singleton attractor); 如果状态数大于一个, 则称为吸引环(cyclic attractor)[20].单吸引子就是在没有基因扰动的条件下, 状态不断更新的过程中, 遇到的某一个无法离开的点.吸引环就是, 在没有基因扰动的条件下, 状态不断更新的过程中, 遇到的某些无法离开的点.
1.2.2 吸引子举例吸引子在生物学中有着非常重要的作用, 在系统生物学中, 有的研究者认为布尔网络的吸引子应该反映细胞的类型, 也有研究者认为吸引子代表了细胞的状态, 如增生、凋亡、分化等[20], 并有研究人员加以了证明[5-6].此处对吸引子举例说明, 见图 1所示.在图 1(a)中, 状态110就是一个吸引子, 因为布尔网络被吸引在110这个状态无法去别的状态了, 并且它是单个的点, 所以这是单吸引子; 在图 1(b)中,
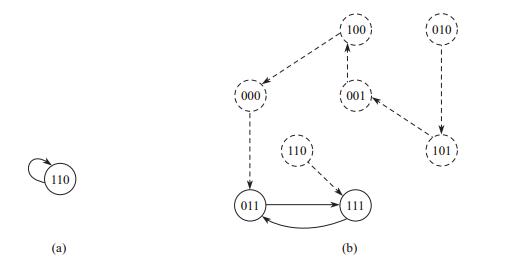
|
图 1 a) 布尔网络单吸引子举例; (b)布尔网络吸引环举例 Fig.1 (a) An example of Boolean network singleton attractors; (b) An example of Boolean network cyclic attractors |
虽然, 布尔网络移动到吸引子中之后无法继续移动, 但是这只是理想情况, 在实际中, 存在一些别的因素会影响布尔网络的状态.众所周知, 因为各种物理、化学因素, 基因存在小概率的基因突变, 也会因为外界因素由激活变为抑制, 或者由抑制变为激活, 这种情况被称为基因扰动.布尔网络也对这样的情况进行了定义, 加入了扰动函数.本文涉及的基因扰动可以分为3种[22], 分别是基因淘汰、基因过度表达、基因交互淘汰.
基因淘汰表示在布尔网络转移的过程中, 基因会有一定概率置0, 也就是由激活转换为抑制, 而这个抑制过程与该点正常状况下的布尔更新函数无关.基因过度表达与元素淘汰相反, 表示在布尔网络转移的过程中, 基因会有一定概率置1, 也就是由抑制转换为激活.同样, 这个激活过程与该点正常状况下的布尔更新函数无关.基因交互淘汰是指, 把影响某基因的一些基因置0, 例如基因
PRISM[1]是由伯明翰大学和牛津大学联合开发的一款模型验证工具, 它的主要功能之一是用于建模和分析具有随机性行为的系统. PRISM支持3种不同的模型, 即离散时间马尔科夫链(DTMC)、连续时间马尔科夫链(CTMC)、马尔科夫决策过程(MDP).
1.3.1 基本语法在PRISM中, 一个模型是由很多个包含变量的并且能够互相交互的模块组成的.变量在某一给定时刻的取值构成了该模块的状态, 每一个模块的状态共同决定了整个模型的状态.模块的声明方式是
module modu_name
vari_name:[min..max] init init_num;
endmodule,
其中, 模块名称为modu_name, 模块中有名为vari_name的变量, 后而跟着的是表示变量范围的区间, 以及可选的初始值参数.
模块的行为, 即它的状态转换, 是由一系列带有守卫条件(guard)的状态转换函数来指定的, 状态转换函数的格式为
| $ \text{ }\!\![\!\!\text{ action }\!\!]\!\!\text{ guard- > prob }\!\!\_\!\!\text{ 1:update }\!\!\_\!\!\text{ 1+prob }\!\!\_\!\!\text{ 2:update }\!\!\_\!\!\text{ 2+L,} $ | (2) |
其中, 行为(action)是一个进程代数形式的行为标签, 它将同步的机制带入到模型中.在一个DTMC模型中, 如果要触发一个带有行为标签的状态转换函数, 那么要同步地检查其他具有相同行为标签的状态转换函数的守卫条件, 如果所有相同行为标签的状态转换函数的守卫条件都满足, 那么所有相同行为标签的状态转换函数将会同步执行, 只要其中有一个守卫条件不满足, 那么所有的相同行为标签的状态转换函数都将不会被执行.
1.3.2 回报信息PRISM模型可以扩充入回报信息, 可以分析与所给汇报的期望值有关的属性. PRISM中的DTMC可以扩充两种类型的回报, 分别是
状态回报:根据处于某一状态的时间来累加计算的回报;
转换回报:根据某一转换发生的次数来累加计算的回报.
PRISM中回报的结构定义是
rewards "rewardname"
它包含了一个或者多个状态回报项目, 即
guard: reward,
[act]guard: reward.
上述结构中的守卫(guard)与上一小节基本语法中的guard一样, 行为标签(act)是出现在模型的命令中的标签, 回报(reward)是一个具有实际值的表达式.
1.3.3 连续统计逻辑PCTL在PRISM中验证性质时, 需要使用概率时序逻辑PCTL来描述性质, 其定义如下.
定义3 概率时序逻辑PCTL
| $ \begin{align} \phi ::=\text {true}|a|\neg \phi|\phi\wedge\phi|P_{\sim p}[\phi U^I\phi]|S_{\sim p}[\phi], \end{align} $ | (3) |
其中,
如果一个状态
| $ \begin{align} P_{ < 0.15}[\phi_2U\phi_2]. \end{align} $ | (4) |
公式(4)的含义为判断
在本文中, 主要使用到的公式是
| $ \begin{align} S_{=?}[\phi=1], \end{align} $ | (5) |
| $ \begin{align} P_{=?}[F[T, T]\phi=1], \end{align} $ | (6) |
| $ {{R}_{\{"\phi \text{active"}\}=?}}[C\le T],\quad \text{rewards"}\phi \text{active"},\quad \phi \text{=1}:\text{1}, $ | (7) |
其中,
本节将提出一个布尔网络转换为离散时间马尔科夫链的方法, 将转换后的DTMC写入PRISM中, 最后通过验证方法找出原布尔网络吸引子与基因间相互关系.
2.1 总技术路线生物中寻找吸引子的问题, 可以看成是模型检测的形式化问题, 更进一步地, 可以通过在布尔网络中验证基因长期激活概率来找到吸引子.布尔网络中的吸引子分为单吸引子和吸引环, 单吸引子是指所有基因经过一定时间后, 激活抑制情况不变; 吸引环是指一定时间后, 某些基因稳定地轮流激活, 其他基因激活抑制情况不变.在PRISM中, 通过使用DTMC和PCTL可以验证系统的如下性质.
(1) 状态在较长时段内为1的概率.即在一段较长的时间后, 状态(基因)为1(激活)的概率.
(2) 状态在某时刻为1的概率.即在某一时刻, 状态(基因)为1(激活)的概率.
因此, 这两个性质的验证组合起来, 就可以得知基因经过一段时间后激活抑制的情况, 也可以得知基因在不同时刻的激活概率变化情况, 从而计算出该布尔网络的单吸引子和吸引环.这样, 就找到了布尔网络中的单吸引子、吸引环, 解决了吸引子找寻的问题.
本文使用添加基因扰动的方法, 来寻找基因之间的相互关系.基因扰动包括基因抑制、基因过度表达.对某个基因实行基因抑制, 指的是该基因的激活概率降低.类似地, 对某个基因实行基因过度表达, 指的是该基因激活概率增加.在PRISM中, 实现基因抑制与基因过度表达可以通过改变激活的概率来实现, 而加入基因扰动后的结果, 即其他基因会受到怎样的影响, 则可以用PRISM的回报信息来记录.举个例子, 如果在某基因调控网络中加入基因
综上所述, 我们通过对基因调控网络使用DTMC建模, 并使用PRISM的统计概率验证方法, 就能够找到吸引子(单吸引子、吸引环)和基因间的相互关系.
图 2展示了总体的技术路线.要完成吸引子和基因间相互关系的找寻需要经过若干步骤.首先, 根据相关信息选定该布尔网络的初始状态; 然后将更新函数通过真值表转换的方式转换为DTMC, 写入PRISM; 最后, 在PRISM中通过模型验证的方法计算吸引子, 通过基因扰动的方法计算基因间相互关系.
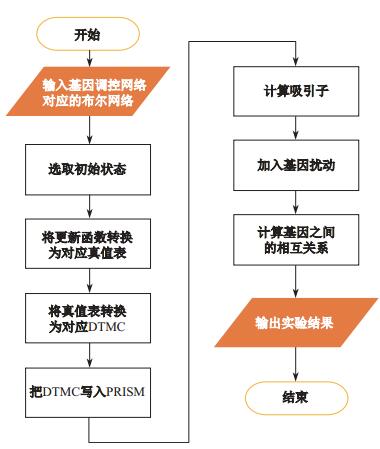
|
图 2 总技术路线 Fig.2 The overall technical route |
将布尔网络转换为DTMC模型, 首先要将布尔网络的更新函数转换为对应的真值表, 因为在布尔网络中, 更新函数表达了布尔网络的变化过程, 包含了布尔网络的主要信息, 所以转换布尔网络只需要研究如何转换更新函数即可.将布尔网络转换为DTMC模型的技术路线如图 3所示.首先输入基因调控网络对应的布尔网络; 然后将布尔网络的更新函数转换为真值表, 对每个基因建立一个DTMC模型; 再后找到真值表中涉及的该基因条目, 写入其DTMC的概率转移矩阵中, 在不同基因的DTMC模型之间, 通过PRISM中的标签使得它们的状态转移同步; 最后输出所有基因对应的DTMC.
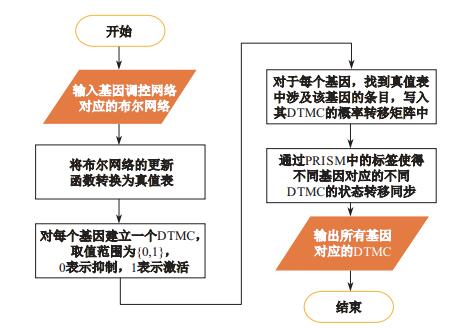
|
图 3 布尔网络转换为DTMC模型技术路线 Fig.3 The transition technical route from Boolean network to DTMC |
以EKLF更新函数为例[22], EKLF=Gata1
| 表 1 EKLF的更新函数转换产生的真值表 Tab.1 The truth table of gene EKLF's update function |
用布尔更新函数的真值表便于分析, 即使在更新函数复杂的情况下, 也可以直接得到DTMC模型的状态转换函数.接下来可以将真值表形式的布尔网络转换为DTMC模型, 并直接以PRISM的语法形式写出.首先, 把每个基因当作是一个模块, 所有的基因共同构成一个DTMC模型, 每个基因对应的变量可以取0或者1两种取值, 分别表示该基因未表达和表达, 即抑制和激活状态.所以EKLF、Gata1和Fli1的声明如下.
module Gata1
Gata1:[0..1] init 0;
endmodule
module EKLF
EKLF:[0..1] init 0;
endmodule
module Fli1
Fli1:[0..1] init 0;
endmodule
状态更新函数表示状态的变化情况, 写出EKLF的PRISM更新函数需要逐行改写EKLF的真值表.例如第一条, Gata1和Fli1取值为0时, 计算得知EKLF取值也为0的内容就可以按照PRISM的基本语法写作.
[areklf_1]Gata1=0
[areklf_1]Fli1=0
[areklf_1]EKLF=1
由于每个模块内部只能修改自己模块内部的变量, 所以需要把以上代码块中的3条语句分别放在对应的3个模块内部, 然后用守卫条件来同步执行.这样, 只有3条语句的守卫条件同时满足时, 才会执行这一条更新语句.类似, EKLF真值表中后3条更新语句的状态更新函数如下.
[areklf_2]Gata1=0
[areklf_2]Fli1=1
[areklf_2]EKLF=1
[areklf]Gata1=1
[areklf]Fli1=0
[areklf]EKLF=0
[areklf_3]Gata1=1
[areklf_3]Fli1=1
[areklf_3]EKLF=1 -
每一条语句开头方括号里的守卫表示同步执行, 需要同样守卫语句的所有条件同时满足, 才会同时执行这些语句.然后需要将这些语句分别写入对应模块中.最终的转换为DTMC的PRISM代码如下所示.
module Gata1
Gata1:[0..1] init 0;
[areklf_1]Gata1=0
[areklf_2]Gata1=0
[areklf]Gata1=1
[areklf_3]Gata1=1
endmodule
module Fli1
Fli1:[0..1] init 0;
[areklf_1]Fli1=0
[areklf_2]Fli1=1
[areklf]Fli1=0
[areklf_3]Fli1=1
endmodule
module EKLF
EKLF:[0..1] init 0;
[areklf_1]EKLF=1
[areklf_2]EKLF=1
[areklf]EKLF=0 -
[areklf_3]EKLF=1
endmodule.
至此, 就把EKLF的更新函数Gata1
而要找到基因之间的相互关系, 则需要加入基因扰动.基因扰动是指基因在受到某些外界因素比如物理因素、化学因素干扰之后, 有一定概率由激活转换为抑制, 或者由抑制转换为激活的效果, 而这些转换与原本的基因更新无关.在第1节中已经提到有基因淘汰、基因过度表达和基因交互淘汰[22]3种主要的扰动方式.这里, 本文加入基因淘汰和基因过度表达, 分别表示基因由激活变为抑制、基因由抑制变为激活.仍以EKLF基因为例, 如果要加入基因淘汰, 假设这个扰动发生的概率为
module EKLF
EKLF:[0..1] init 0;
[areklf_1]EKLF=1 -
[areklf_2]EKLF=1 -
[areklf]EKLF=0 -
[areklf_3]EKLF=1 -
endmodule.
即当基因有概率
module EKLF
EKLF:[0..1] init 0;
[areklf_1]EKLF=1 -
[areklf_2]EKLF=1 -
[areklf]EKLF=0 -
[areklf_3]EKLF=1 -
endmodule.
EKLF的代码中有3条是激活状态, 在有
根据以上方法, 就可以将基因调控网络的布尔网络转换为DTMC, 然后写入模型检测工具PRISM中.接下来介绍如何使用PRISM找到吸引子、如何通过基因扰动找到基因之间相互关系.
2.3 找吸引子、基因之间相互关系的方法布尔网络中的吸引子分为单吸引子和吸引环, 前者所表现出来的情况是所有基因激活抑制情况不变, 后者所表现出来的情况是长期来看, 某些基因轮流激活, 其他基因激活抑制情况不变.在PRISM中, 可以计算某基因长期激活的概率与在未来某个时间点激活的概率, 它们的公式分别是
| $ \begin{align} S_{=?}[\text {EKLF}=1], \end{align} $ | (8) |
| $ \begin{align} P_{=?}[F[T, T]\text {EKLF}=1]. \end{align} $ | (9) |
公式(8)表示EKLF基因长期激活的概率, 式(9)表示EKLF基因在时间点
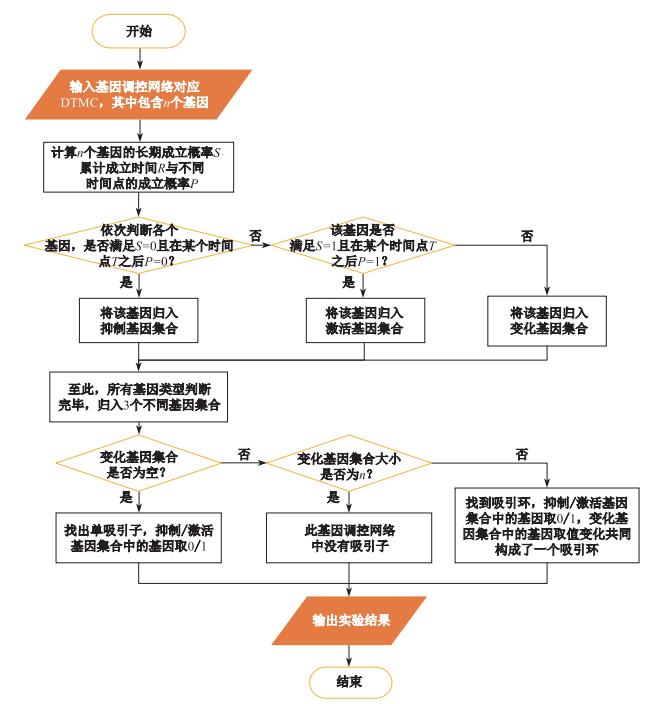
|
图 4 吸引子找寻技术路线 Fig.4 The technical route of detecting attractors |
目前对于吸引子找寻的研究通常分开来解决单吸引子与吸引环的找寻问题, 且通常方法较为复杂.传统算法有Zhang等人提出的一系列基于迭代扩展局部基因表达谱的单吸引子找寻算法[23].该系列算法需要读取所有已知的基因状态是激活还是抑制, 建立已知基因的基因表达谱, 然后对于每个已知基因, 进行逐个基因的扩展, 判断当前局部已知基因表达谱是否仍然是一个单吸引子, 如果是, 则继续扩展下一基因, 直到无法再扩展; 如果不是则结束当前基因表达谱的分支测试, 进入下一个已知基因的表达谱分支测试.本文利用形式化方法工具PRISM可以直接算出每个基因长期成立概率与各时间点成立概率的特点, 简化了寻找吸引子的流程, 使之不必从每个基因的激活抑制情况扩展来寻找吸引子, 且此方法可以同时找出单吸引子与吸引环, 或是判断不存在吸引子, 而不必使用多个算法来分别计算; 并且, 在此方法的基础上还可以通过增加基因扰动, 使用形式化方法工具PRISM直接计算出每个基因累计成立概率与长期成立概率的特点来找出基因间相互关系, 加深对已建立的基因调控网络的理解.
对于基因之间的相互关系, 使用上一小节方法加入基因扰动后, 可以使用长期激活概率和PRISM中的回报信息来验证基因扰动后对其他基因的影响. PRISM中回报信息用于计算某基因截止到某一时刻为激活状态的时间之和.计算EKLF基因在
| $ \begin{align} R_{\{"\text {EKLFactive}"\}=?}[C\leq T], \quad \text {rewards"EKLFactive"}, \quad \text {EKLF}=1:1. \end{align} $ | (10) |
如果加入某基因的抑制扰动, 其他基因长期激活概率或者累计激活时间提高, 那么就说明这个基因对其他基因有着激活的作用, 反之亦然.这样就可以找到各个基因之间的相互促进或是抑制的关系, 即便在原本的布尔网络中, 难以发现的促进或抑制关系, 找到基因之间的相互关系有助于找到某些疾病的治疗方向.寻找基因间相互关系的技术路线如图 5所示.
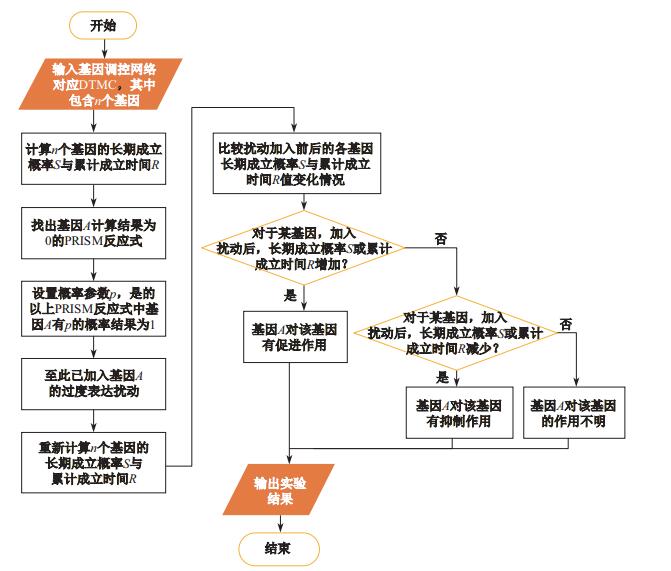
|
图 5 基因间相互关系找寻技术路线 Fig.5 The technical route of detecting gene relations |
目前对于基因间相互关系的研究主要有: ①通过聚类分析找到基因间相互关系, 主要是对采自微阵列的数据进行聚类分析基因表达谱, 辨明基因表达模式[24]; ②进行反复的微扰分析找出基因间相互关系[25]; ③通过"反向技术"或"逆向工程"来推断网络, "反向技术"就是从基因表达的数据反向推断未知的或隐含的基因间相互关系结构的技术, 它需要选定合适的参数模型, 并用适当的算法推断网络参数, 确定输入输出规则, 预测在不同时间上基因间相互关系的变化[26].这些方法都是在基因实验数据的基础上找基因间相互关系, 而本文则是在已有的基因调控网络中挖掘基因间相互关系, 相比较于以上所述方法, 由图 5中的找寻方法流程可知, 本文方法更为简单易用:在建立了DTMC模型的基础上, 只需要通过设置扰动概率参数增加基因扰动, 就可以使用形式化方法工具PRISM直接计算出每个基因累计成立概率与长期成立概率, 以此直接找出原本隐藏的基因间相互关系, 加深对已建立的基因调控网络的理解.并且步骤清晰, 易用.综上所述, 本文以一种简洁的形式化方法将基因调控网络转换为离散时间马尔科夫链, 解决了寻找单吸引子、寻找吸引环、寻找基因间相互关系这3个问题, 得到了有效的成果.
3 大鼠干细胞基因调控网络验证与分析本节以大鼠干细胞基因调控网络的布尔网络[27]为例, 介绍布尔网络到离散时间马尔科夫链的整个转换过程, 以及转换结束后写入PRISM进行吸引子查找实验, 加入基因扰动进行基因之间相互关系实验, 最后得出实验结果并加以分析.大鼠干细胞基因调控网络是一个有着著名生物学背景和先进实验研究结果的系统, 它被生物界广泛地认为是干细胞生物学中的一个经典范例[28].
3.1 吸引子寻找方法与分析大鼠干细胞基因调控网络的布尔网络更新函数如表 2所示.
| 表 2 大鼠干细胞基因布尔更新函数 Tab.2 Mice stem cell gene regulatory network's Boolean update function |
使用第1节的方法将此布尔网络的每一条更新函数依次转换为离散时间马尔科夫链并写入PRISM中.将布尔网络转换为离散时间马尔科夫链后, 依据文献[22], 选取一种初始状态Gata2、Cebpa、Pu.1激活, 其余基因抑制作为本实验的初始状态.这样设置之后, 模型的计算结果如表 3所示.
| 表 3 吸引子找寻相关信息计算结果 Tab.3 The result of attractor finding information |
由表 3可得基因Gata2、Gata1、Fog1、EKLF、Fli1以及Scl的长期激活概率都为0, 同时它们在某一时刻之后的激活概率都为0, 因此可以判断它们属于抑制基因集合; 并且可以看出, 基因Cebpa和Pu.1的长期激活概率都为1, 且在某一时间点后的成立概率为1, 所以可以判断它们属于激活基因集合.最后, 剩余基因cJun、EgrNab和Gfi1都属于变化基因集合.根据图 4的吸引子判断流程可以得知, 此基因调控网络存在一个吸引环.综上所述, 由表 3中数据可以得出, 在Gata2、Cebpa、Pu.1激活, 其余基因抑制的初始状态下, 这个基因调控网络会被吸引到cJun、EgrNab和Gfi1这3个基因上, 它们的激活-非激活状态根据更新函数的变化情况共同构成了一个吸引环.由上文所述, 在生物学的基因调控网络中, 吸引环是研究细胞系统状态的关键, 它对应于细胞系统的特定稳定状态.与传统方法相比较, 本文方法的更为简单易用, 利用了形式化方法工具PRISM可以直接算出每个基因长期成立概率与各时间点成立概率的特点, 简化了寻找吸引子的流程, 使之不必从每个基因的激活抑制情况扩展来寻找吸引子, 且此方法可以同时找出单吸引子与吸引环, 或是判断不存在吸引子, 而不必使用多个算法来分别计算.
3.2 基因促进/抑制相互关系的分析结果与验证在找到吸引环之后, 为了更为贴近实际实验的结果, 对各个基因加入基因淘汰和基因过度表达的扰动, 其中, 在对Gata1进行基因过度表达扰动时, 发现了原布尔网络中不易看出的基因之间促进抑制的关系. Gata1的基因过度表达扰动结果见表 4.
| 表 4 基因Gata1的过度表达扰动结果 Tab.4 The result of gene Gata1's over expression |
表 4为加入Gata1的基因过度表达扰动后的数据计算结果, 表中, 用红色代表加入扰动之后相关数值上升, 蓝色代表加入扰动之后相关数值下降, 可以通过这些数据找出Gata1与其他基因的相互关系.加入基因过度表达扰动也就是对于每个Gata1基因反应结果为抑制的更新函数, 增加概率参数使得Gata1基因在这个更新函数下可以概率地为激活, 这个概率参数就是Gata1基因被扰动的概率.其实际意义为, Gata1基因在每次更新状态时, 存在这样一个被扰动的概率.本文将这个概率设置为0.5, 这样设置之后的验证结果如最右一列所示.可以看出, 加入了Gata1基因过度表达扰动之后, 基因Gata1、Fog1、EKLF、cJun、EgrNab的
因此, 本文试验方法可以直接看出原本布尔网络中隐藏着的基因之间的促进、抑制关系, 而已有文献证明, Gfi1的表达不足大鼠会罹患白血球减少症[29].本实验中得出Gata1的表达对Gfi1是有着抑制作用的这一结论, 就可以提供一个白血球减少症的治疗思路.类似, 找出这种隐藏着的基因促进/抑制关系对于解决人类的疾病也有着积极的作用.因此本文实验的方法是有意义的.相比较于传统方法, 本文方法更为简单易用, 在建立的DTMC模型的基础上, 只需要通过设置扰动概率参数增加基因扰动, 就可以使用形式化方法工具PRISM直接计算出每个基因累计成立概率与长期成立概率, 以此直接找出原本隐藏的基因间相互关系, 加深对已建立的基因调控网络的理解.
4 总结和展望 4.1 总结为了解决生物工程中基因调控网络难以找吸引子这一重大问题, 本文提出了使用形式化方法中概率模型检测来探查吸引子的方法:首先将布尔网络表示的基因调控网络的更新函数表示为对应的真值表; 然后将真值表转换为离散时间马尔科夫链, 写入模型检测工具PRISM中; 再后通过PRISM中的"长期是否成立"性质检测每个基因在很长一段时间之后的激活概率, 以此找到吸引子.经检测, 大鼠干细胞基因中有5个基因会在一段时间后激活, 它们共同构成了一个吸引环.整个检测流程简洁易用, 有着直接找出吸引子的效果.同时, 为了解决生物工程中基因之间隐藏的相互促进/抑制关系不易寻找的问题, 本文提出了使用形式化方法中的概率模型检测的方法来寻找基因之间相互关系的方法, 通过添加基因扰动, 改变每个基因的激活/抑制概率, 找到每个基因对其他基因的促进/抑制关系.经实验, 找出了大鼠干细胞中Gata1基因的抑制/促进对象, 此实验结果对解决大鼠的白血球减少症有着治疗方面的意义.综上所述, 本文以一种简洁的形式化方法将基因调控网络转换为离散时间马尔科夫链并解决了寻找单吸引子、寻找吸引环、寻找基因间相互关系这3个问题, 得到了有效的成果.
4.2 展望布尔网络中寻找吸引子最大的问题在于时间复杂度, 本文并未涉及这个问题, 因而, 下一步的工作就在于在本文转换为离散事件马尔科夫链的基础上, 如何降低转换算法的时间复杂度, 进而降低整体在布尔网络中寻找吸引子问题的时间复杂度, 从而在简洁流程的基础上, 将此方案用在更为广阔的实际场景上.
| [1] | KWIATKOWSKA M, NORMAN G, PARKER D. PRISM:Probabilistic symbolic model checker[C]//International Conference on Computer Performance Evaluation. Berlin:Springer, 2002:200-204. |
| [2] | KAUFFMAN S. Homeostasis and differentiation in random genetic control networks[J]. Nature, 1969, 5215(224): 177-178. |
| [3] | TRAIRATPHISAN P, MIZERA A, PANG J, et al. Recent development and biomedical applications of probabilistic Boolean networks[J]. Cell Communication and Signaling, 2013, 11: 46 DOI:10.1186/1478-811X-11-46 |
| [4] | AKUTSU T, MELKMAN A A, TAMURA T, et al. Determining a singleton attractor of a Boolean network with nested canalyzing functions[J]. Journal of Computational Biology, 2011, 18(10): 1275-1290. DOI:10.1089/cmb.2010.0281 |
| [5] | HUANG S. Gene expression profiling, genetic networks, and cellular states:An integrating concept for tumorigenesis and drug discovery[J]. Journal of Molecular Medicine, 1999, 77(6): 469-480. DOI:10.1007/s001099900023 |
| [6] | HUANG S. Genomics, complexity and drug discovery:Insights from Boolean network models of cellular regulation[J]. Pharmacogenomics, 2001, 2(3): 203-222. DOI:10.1517/14622416.2.3.203 |
| [7] | DROSSEL B, MIHALJEV T, GREIL F. Number and length of attractors in a critical Kauffman model with connectivity one[J]. Physical Review Letters, 2005, 94(8): 088701 DOI:10.1103/PhysRevLett.94.088701 |
| [8] | KAUFFMAN S A. The origins of order:Self-organization and selection in evolution[J]. Journal of Evolutionary Biology, 1992, 13(1): 133-144. |
| [9] | SAMUELSSON B, TROEIN C. Superpolynomial growth in the number of attractors in Kauffman networks[J]. Physical Review Letters, 2003, 90(9): 098701 DOI:10.1103/PhysRevLett.90.098701 |
| [10] | TAMURA T, AKUTSU T. An O(1.787n)-time algorithm for detecting a singleton attractor in a Boolean network consisting of AND/OR nodes[C]//Fundamentals of Computation Theory, 16th International Symposium, FCT 2007, Proceedings. 2007:494-505. |
| [11] | ZHENG Q B, SHEN L Z, SHANG X Q, et al. Detecting small attractors of large Boolean networks by functionreduction-based strategy[J]. IET Systems Biology, 2016, 10(2): 49-56. DOI:10.1049/iet-syb.2015.0027 |
| [12] | HE Q B, XIA Z L, LIN B. An efficient approach of attractor calculation for large-scale Boolean gene regulatory networks[J]. Journal of Theoretical Biology, 2016, 408: 137-144. DOI:10.1016/j.jtbi.2016.08.006 |
| [13] | MEYN S P, TWEEDIE R L. Markov Chains and Stochastic Stability[M]. Berlin: Springer Science & Business Media, 2012. |
| [14] | HOSOYA K, TOMI M, OHTSUKI S, et al. Enhancement of L-cystine transport activity and its relation to xCT gene induction at the blood-brain barrier by diethyl maleate treatment[J]. The Journal of Pharmacology and Experimental Therapeutics, 2002, 302(1): 225-231. DOI:10.1124/jpet.302.1.225 |
| [15] | LI G, ROSS K E, ARIGHI C N, et al. MiRTex:A text mining system for miRNA-gene relation extraction[J]. PLOS Computational Biology, 2015, 11(9): e1004391 DOI:10.1371/journal.pcbi.1004391 |
| [16] | 朱祥, 张云秋, 冯佳. 基于生物医学文本挖掘工具的白血病和基因关系研究[J]. 中华医学图书情报杂志, 2015, 24(10): 28-32. DOI:10.3969/j.issn.1671-3982.2015.10.007 |
| [17] | HOWARD R A. Dynamic Probabilistic Systems[M]. New York:Dover Publicatinos Inc, 2007. |
| [18] | KWIATKOWSKA M, NORMAN G, PARKER D. Advances and challenges of probabilistic model checking[C]//Communication, Control, and Computing. IEEE, 2010:1691-1698. |
| [19] | LIANG S D, FUHRMAN S, SOMOGYI R. Reveal, a general reverse engineering algorithm for inference of genetic network architectures[J]. Pacific Symposium on Biocomputing, 1998(3): 18-29. |
| [20] | 郑启奔. 布尔网络吸引子确定算法研究[D]. 浙江温州: 温州大学, 2015. |
| [21] | TAMURA T, AKUTSU T. Detecting a singleton attractor in a Boolean network utilizing SAT algorithms[J]. Ieice Trans Fundamentals, 2009, 92(2): 493-501. |
| [22] | KRUMSIEK J, MARR C, SCHROEDER T, et al. Hierarchical differentiation of myeloid progenitors is encoded in the transcription factor network[J/OL]. PlOS One, (2011-08-10)[2016-08-21]. https://doi.org/10.1371/journal.pone.0022649. |
| [23] | ZHANG S Q, HAYASHIDA M, AKUTSU T, et al. Algorithms for finding small attractors in Boolean networks[J]. Eurasip Journal on Bioinformatics and Systems Biology, 2007: 20180 DOI:10.1155/2007/20180 |
| [24] | LEE W P, HSIAO Y T. An adaptive GA-PSO approach with gene clustering to infer S-system models of gene regulatory networks[J]. Computer Journal, 2011, 54(9): 1449-1464. DOI:10.1093/comjnl/bxr038 |
| [25] | SHLYKOVA I, PONOSOV A. Singular perturbation analysis and gene regulatory networks with delay[J]. Nonlinear Analysis:Theory Methods & Applications, 2010, 72(9/10): 3786-3812. |
| [26] | NICHOLSON C, GOODWIN L, CLARK C. Variable neighborhood search for reverse engineering of gene regulatory networks[J]. Journal of Biomedical Informatics, 2017, 65: 120-131. DOI:10.1016/j.jbi.2016.11.010 |
| [27] | FISHER J, KÖKSAL A S, PITERMAN N, et al. Synthesising executable gene regulatory networks from singlecell gene expression data[C]//International Conference on Computer Aided Verification. Springer International Publishing, 2015:544-560. |
| [28] | ORKIN S H, ZON L I. Hematopoiesis:an evolving paradigm for stem cell biology[J]. Cell, 2008, 132(4): 631-644. DOI:10.1016/j.cell.2008.01.025 |
| [29] | KARSUNKY H, ZENG H, SCHMIDT T, et al. Inflammatory reactions and severe neutropenia in mice lacking the transcriptional repressor Gfi1[J]. Nature Genetics, 2002, 30(3): 295-300. DOI:10.1038/ng831 |




